-
Notifications
You must be signed in to change notification settings - Fork 10
/
Copy pathch16.Rmd
123 lines (96 loc) · 2.92 KB
/
ch16.Rmd
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
---
title: "分群模型(Clustering)"
author: "郭耀仁"
date: "`r Sys.Date()`"
output: slidy_presentation
---
```{r setup, include=FALSE}
knitr::opts_chunk$set(echo = TRUE, warning = FALSE, results = 'hide', message = FALSE)
```
## 常見的機器學習問題
- 監督式學習
- 迴歸模型
- 分類模型
- 非監督式學習
- 分群模型(*)
## 分群模型的特性
- 訓練資料集是沒有答案的, 所以沒有對與錯
- 組內差異小
- 組間差異大
- 差異是以觀測值之間的**距離**作為度量
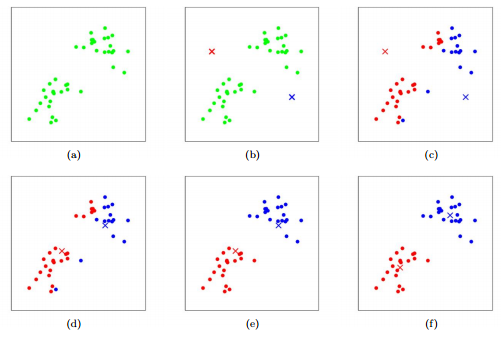
## K-Means
- 隨便放 k 個中心點
- 依照各點與 k 個中心點的距離標記分群
- 然後將中心點移到各群的中心
- 重複上兩個步驟一直到中心點的移動已經收斂
- 如下圖(k = 2):
## `kmeans()` 函數
- 查詢 `kmeans()` 函數
- `centers` 就是 k 值
- `nstart` 是指重新隨意放 k 個中心點的次數, 一般建議 `nstart >= 10`
## `kmeans()` 函數(2)
```{r}
iris_km <- iris[, -5]
set.seed(87)
km_fit <- kmeans(iris_km, nstart = 10, centers = 3)
# Plotting
par(mfrow = c(1, 2))
plot(x = iris$Sepal.Length, y = iris$Sepal.Width, col = iris$Species, main = "Labeled")
plot(x = iris$Sepal.Length, y = iris$Sepal.Width, col = km_fit$cluster, main = "K-Means", ylab = "")
```
## 評估分群模型
- 組內差異(WSS)愈小愈好
- 組間差異(BSS)愈大愈好
- 比例 `Total WSS/Total SS`,這個數值愈低表示績效愈好
```{r}
ratio <- km_fit$tot.withinss / km_fit$totss
ratio
```
## 選擇 k 值
- 隨著 k 增加, `Total WSS/Total SS` 一定會持續下降
- 如果我們讓 k = 觀測值數目, 一定可以得到一個 `Total WSS/Total SS` 最低的結果
- 但這個不會是我們想要的分群方法
- 我們要找的是下降效率開始變小的 k
- 實務上常使用**陡坡圖**來找**手肘點**
## 選擇 k 值(2)
```{r}
ratio_ss <- rep(NA, times = 20)
for (k in 1:length(ratio_ss)) {
iris_km <- iris[, -5]
set.seed(87)
km_fit <- kmeans(iris_km, centers = k, nstart = 10)
ratio_ss[k] <- km_fit$tot.withinss/km_fit$totss
}
plot(ratio_ss, type = "b", xlab = "k", main = "screeplot")
```
## 標準化
- Standard Scaler
- 使用 `scale()` 函數
```{r}
scaled_iris_mat <- scale(iris[, -5])
scaled_iris_df <- data.frame(scaled_iris_mat)
View(scaled_iris_df)
```
## 標準化(2)
- Min Max Scaler
- 自訂
```{r}
# Self-defined function: min_max_scaler
min_max_scaler <- function(df) {
col_n <- ncol(df)
empty_matrix <- matrix(nrow = nrow(df), ncol = ncol(df))
normalized_df <- data.frame(empty_matrix)
for (i in 1:col_n) {
minimum <- min(df[, i])
maximum <- max(df[, i])
normalized <- (df[, i] - minimum) / (maximum - minimum)
normalized_df[, i] <- normalized
}
names(normalized_df) <- names(df)
return(normalized_df)
}
# Function calls
scaled_iris <- min_max_scaler(iris[, -5])
View(scaled_iris)
```